speciesTag - Extraction de noms d’espèces
Ce service web détecte dans un texte les noms scientifiques d’espèces animales, végétales (ainsi que les virus, bactéries, champignons,
chromistes, protistes, etc.).
Ce service fonctionne quelle que soit la langue à condition qu’elle soit dans un alphabet latin.
Ce web service, appelé speciesTag ou IRC3sp, est une version de l’outil IRC3 dédiée à la recherche des noms scientifiques — ou noms binominaux — d’espèces animales, végétales ou autres dans un corpus de textes en se référant à une liste finie (mais, aussi exhaustive que possible).
Pour mémoire, en taxonomie, un nom binominal est formé de deux noms latins (ou latinisés) comprenant le nom de genre et le nom spécifique, comme “Canis lupus” pour le loup. Ce nom est normalement écrit en italique avec une initiale en majuscule pour le nom de genre et il peut être présent sous une forme abrégée où seule l’initiale du nom de genre est indiquée, comme “C. lupus”. À l’exception d’espèces très connues comme Escherichia coli qui est souvent simplement écrit E. coli, la forme abrégée ne doit être utilisée que si la forme longue est déjà apparue au moins une fois. De plus, si un nom de genre a été cité, toutes les espèces appartenant à ce même genre peuvent ensuite être citées sous forme abrégée, comme “Canis lupus, C. latrans et C. aureus” (pour “Canis lupus, Canis latrans et Canis aureus”).
Cependant, les formes abrégées peuvent être ambiguës. Par exemples, on a deux espèces de poissons, Cyprinus carpio et Carpiodes carpio, qui ont la même abréviation : C. carpio. Pour éviter les erreurs, IRC3sp commence par faire la liste des noms de genre présents dans le document analysé pour obtenir l’ensemble des espèces correspondantes dans la ressource, et donc, l’ensemble des abréviations possibles. Malgré cela, si une ambiguïté demeure, on considère comme valide le dernier nom de genre cité in extenso avant l’occurrence de la forme abrégée.
Ce service web ne renvoie rien quand le texte ne contient pas de noms d’espèces scientifiques.
- Catalogue of Life contient 3,8 millions de noms d’espèces, sous des formes différentes
- Catalogue of Life : https://www.catalogueoflife.org/
- IRC3 : https://gitbucket.inist.fr/scodex/IRC3/
[{
"id": 1,
"value": "Trophic diversity accumulation curves of (a) Pseudopercis semifasciata, (b) Acanthistius patachonicus and (c) Pinguipes brasilianus. Horizontal lines show Brillouin diversity index (Hz) values (Hz± 0·05 Hz) and the vertical line shows a value n- 2 (n = number of stomachs)."
},{
"id": 2,
"value": "Phasianus colchicus/versicolor: in our study, the best match."
},{
"id": 3,
"value": "short lower jaw in Etheostoma bellator Suttkus"
}, {
"id": 4,
"value": [
"Carnivore diet analysis based on next‐generation sequencing: application to the leopard cat (Prionailurus bengalensis) in Pakistan ",
"The leopard cat (Prionailurus bengalensis) is a small felid (weight 1.7–7.1 kg; Sunquist & Sunquist 2009), with a wide range in Asia (8.66 × 106 km2; Nowell & Jackson 1996). ",
"Muridae (mainly Rattus spp. and Mus spp.) seem to represent the main prey items throughout the leopard cat distribution range, supplemented by a wide variety of other prey including small mammals such as shrews and ground squirrels, birds, reptiles, frogs and fish (Tatara & Doi 1994; Grassman et al. 2005; Austin et al. 2007; Rajaratnam et al. 2007; Watanabe 2009; Fernandez & de Guia 2011). ",
"More recently, Deagle et al. (2009, 2010) investigated the diet of Australian fur seals (Arctocephalus pusillus) and penguins (Eudyptula minor) by combining a blocking oligonucleotide approach with 454 GS‐FLX pyrosequencing technologies. "
]
}]
[ {
"id": 1,
"value": [ "Acanthistius patachonicus", "Pinguipes brasilianus", "Pseudopercis semifasciata" ]
}, {
"id": 2,
"value": [ "Phasianus colchicus" ]
}, {
"id": 3,
"value": [ "Etheostoma bellator" ]
}, {
"id": 4,
"value": [ "Arctocephalus pusillus", "Eudyptula minor", "Prionailurus bengalensis" ]
}]
Vous souhaitez repérer dans votre corpus des espèces animales et végétales ?
En lançant speciesTag sur vos données depuis Lodex, logiciel libre de visualisation, vous obtiendrez la liste des différentes espèces et des représentations graphiques liées.
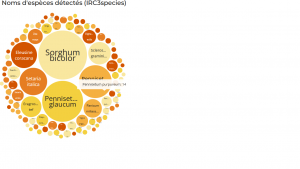
Graphe issu du corpus Istex “Millets“.
Profitez en pour naviguer dans l’ensemble du corpus
et découvrez d’autres corpus scientifiques.


